成績概要書(2004年1月作成)
|
研究課題:イネいもち病真性抵抗性遺伝子Pii に連鎖したDNAマーカーの開発
(ゲノム情報を活用したイネいもち病抵抗性育種技術の開発)
担当部署:中央農業試験場 農産工学部 遺伝子工学科
協力分担:
予算区分:道費(初動研究)
研究期間:2002年度(平成14年度)
|
1.目的
イネゲノム塩基配列データを活用することにより、Pii遺伝子の座乗染色体を決定し、Pii遺伝子と密接に連鎖したDNAマーカーを作出する。
2.方法
供試材料:いもち病真性抵抗性遺伝子型が明らかとされている北海道および本州の品種・系統131種類。
データベース:日本DNAデータバンクDDBJ(DNA Data Bank of Japan)、RGP(Rice
Genome Research Program)
3.成果の概要
1)梶ら(2001)の報告にしたがってRAPDプライマーCA05を用いて、Piiを持つ「石狩白毛」、「藤坂5号」および「ほしのゆめ」のDNAを鋳型にPCRを行い、約1.6 kbの特異断片を得た。
2)特異断片1660 bpの全塩基配列を決定し、日本DNAデータバンクDDBJにアクセスして塩基配列の相同性検索を行った結果、Accesion
number AP005811内の配列に98.288%の相同性で一致し、Pii遺伝子は第9染色体に座乗していることが明らかとなった。
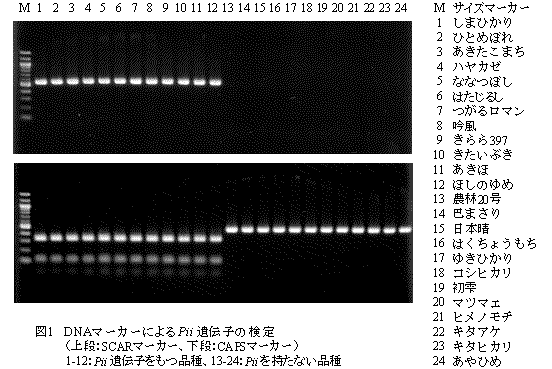
3)特異断片1660bpとAP005811の塩基配列情報を比較解析し、特異的PCRとなるSACR(Sequence Characterized Amplified Regions)マーカーを作成した(図1)。
4)さらに、CAPS(Cleaved Amplified Polymorphic Sequence)マーカーを作成し(図1)、このマーカーをSPI-1と名付けた。CAPSマーカーの優点として以下のことが挙げられる。
(1)DNA精製度の影響が少ないため、簡易抽出法によるDNAでも使用できる。
(2)再現性が高い。
(3)ホモ型とヘテロ型との区別が可能である。
5)北海道および本州品種・系統131種類を用いて、CAPSマーカーによる解析を行った結果、Piiを持つ品種と持たない品種が完全に区別することができ、DNAマーカーによるPii遺伝子の有無の判別が可能となった(表1)。
6)RGPのデータベースからAP005811近傍のクローンの配列を得て、SSR(Simple Sequence
Repeat)配列を探索した。SSRマーカーによる解析により、Pii遺伝子の領域は、第9染色体の26.7〜34.4cM、約1,800kbに絞り込むことができた。
7)イネゲノムデータベースを有効活用することにより、イネいもち病真性抵抗性遺伝子
Piiに連鎖したDNAマーカーSPI-1を極めて短期間に開発できた。SPI-1を利用することにより、育成系統など多数の材料について、
Pii遺伝子の判定を効率良く行うことができ、育種現場で有効である。

4.成果の活用面と留意点
1)水稲の品種・系統におけるPii遺伝子の判定に用いる。
2)遺伝子型の確定には、接種検定を行う必要がある。
5.残された問題とその対応
Pii遺伝子の単離。



